Der Zelloberfläche ein Gesicht geben
ETH-Forscherinnen und -Forscher haben mithilfe von maschinellem Lernen erstmals Proteine auf der Oberfl?che von Zellen umfassend untersucht und katalogisiert. Die Erkenntnisse erm?glichen einen neuen Ansatz in der Medikamentenforschung.

Auf der Oberfl?che von Zellen, verankert in der Zellmembran, befindet sich eine Vielzahl verschiedener Proteine, die für die Zelle lebenswichtige Funktionen ausüben. Sie sind Antennen in die Aussenwelt, die Signale senden und empfangen, dank derer Zellen miteinander kommunizieren k?nnen. Sie sind auch Schleusen für Moleküle und transportieren Stoffe in und aus der Zelle, und sie erm?glichen, dass sich Zellen an anderen Zellen oder Strukturen festhalten k?nnen.
Die Medizin interessiert sich sehr für Oberfl?chenproteine und nutzt sie, um Krankheiten zu behandeln. Rund zwei Drittel der bekannten Medikamente entfalten ihre Wirkung dadurch, dass sie passgenau an ein Oberfl?chenprotein andocken und eine zellul?re Signalkaskade in Gang setzen.
?berholtes Paradigma
Doch so praktisch und einfach das klingen mag: Der bisherige Ein-Medikament-eine-Zielstruktur-Ansatz hat den gewichtigen Nachteil, dass Ziel-Oberfl?chenproteine auch auf vielen unterschiedlichen Zelltypen vorkommen. Viele Medikamente richten sich daher nicht nur an den Zelltyp, den man angreifen m?chte, sondern zus?tzlich auch an andere Zellen. Dies ist eine Ursache, warum viele Medikamente ungewollte Nebenwirkungen hervorrufen.
Forscherinnen und Forscher aus der Gruppe von ETH-Professor Bernd Wollscheid vom Institut für Molekulare Systembiologie und dem Department für Gesundheitswissenschaften und Technologie (D-HEST) erforschen deshalb die Verteilung und Anordnung der Oberfl?chenproteine auf den Zellen. Damit m?chten sie in einem neuen Ansatz geeignetere Ziele für medikament?se Interventionen finden.
Da die Vielfalt der Oberfl?chenproteine von menschlichen Zellen bisher kaum erforscht war, erstellte Wollscheids Doktorandin Damaris Bausch-Fluck zusammen mit dem Bioinformatiker Ulrich Goldmann aus derselben Gruppe in einem ersten Schritt ein Inventar dieser Moleküle. Die Studie wurde vor kurzem in der Fachzeitschrift externe Seite PNAS ver?ffentlicht.
Die Forschenden nutzten dabei die Vorteile maschinellen Lernens: Erstellt wurde das Inventar von einem Computer, dem die Wissenschaftler beibrachten, welche Eigenschaften und Merkmale Oberfl?chenproteine haben. Dazu fütterten sie den Rechner mit zuvor experimentell erhobenen Proteindaten. Dann liessen die Forschenden die Maschine ausrechnen und voraussagen, welche der rund 20'000 Proteine des Menschen an der Oberfl?che von Zellen vorkommen k?nnen.
Weitgehend richtige Voraussagen
Das vom Computer erzeugte Inventar umfasste schliesslich rund 2900 verschiedene Proteine. Mit anderen Worten: Jedes siebte Protein von menschlichen Zellen k?nnte an ihren Oberfl?chen erscheinen. Der neu entwickelte Algorithmus hatte bei seiner Voraussage eine hohe Treffsicherheit. In mehr als 93 Prozent der F?lle lag der Computer richtig, wie eine nachtr?gliche experimentelle ?berprüfung ergab.
Die Zahl von Oberfl?chenproteinen schwankt zudem stark von Zelltyp zu Zelltyp, wie die Forscherinnen zeigen konnten. Die Wissenschaftler benutzten ?ffentlich verfügbare Daten von Zelllinien und konnten zeigen, dass bei Immunzellen nur rund 500 verschiedene Oberfl?chenproteine vorhanden sind, bei Lungen- und Hirnzellen jedoch mehr als 1000. Die gr?sste Vielfalt an Oberfl?chenproteinen besitzen prim?re Stammzellen: 1800 verschiedene Proteine kommen bei ihnen vor. ?Zelllinien haben ein weniger komplexes Oberfl?chenproteom als frisch aus K?rpergewebe isolierten Zellen, da Zelllinien weniger vielf?ltige Wechselwirkungen erfahren?, betont Wollscheid. Die ETH-Forschenden haben ihre Ergebnisse wiederum in einer ?ffentlichen Datenbank hinterlegt.
Organisation und Verteilung entscheidend
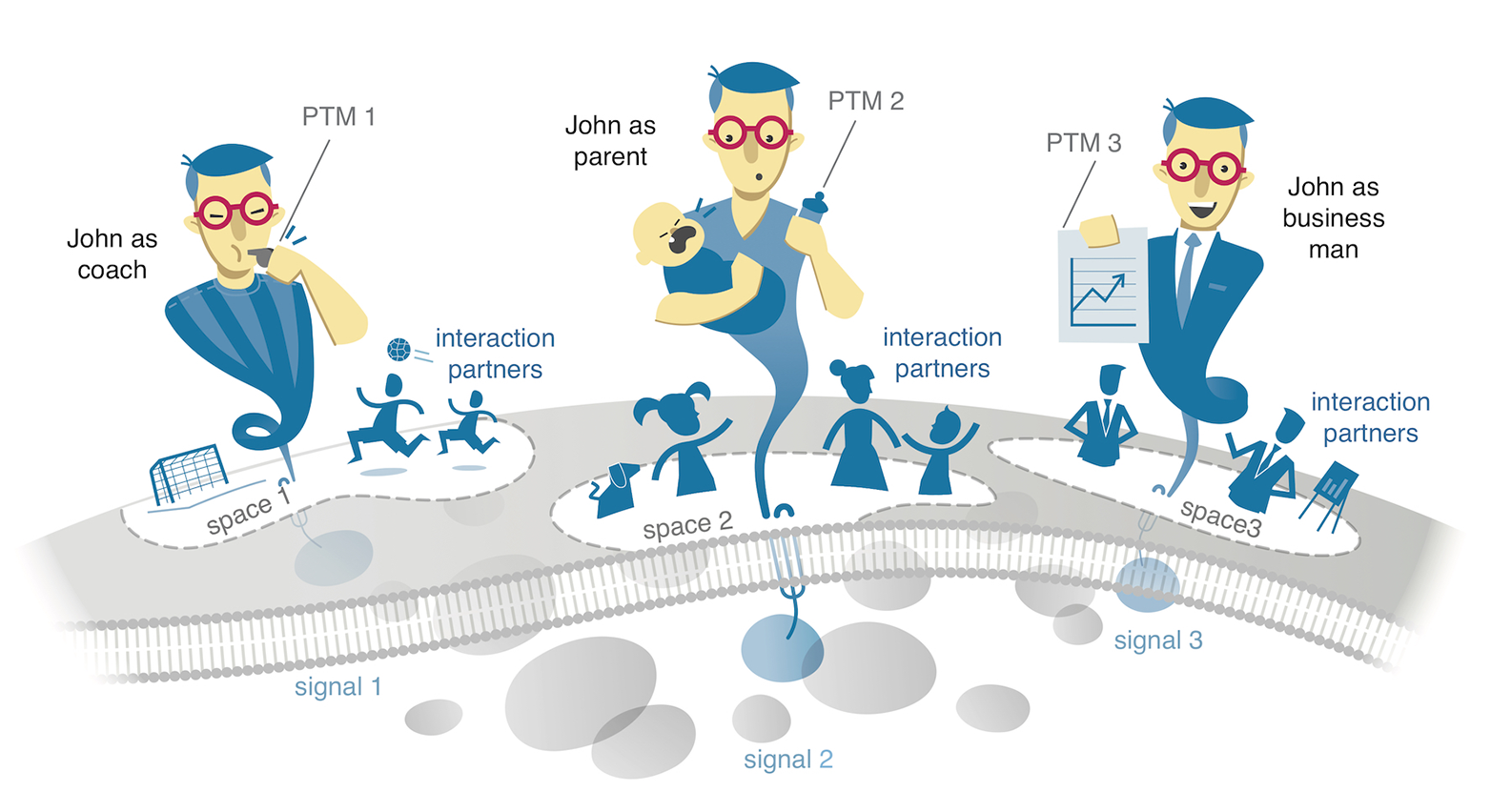
Die Kenntnis über das Vorkommen und die Vielfalt der Oberfl?chenproteine führt allerdings nicht automatisch zu neuen Wirkstoffen. ?Oberfl?chenproteine sind nicht gleichm?ssig verteilt, sondern inselartig angeordnet in funktionalen Einheiten mit wechselnden Proteinzusammensetzungen und somit wechselnden Funktionen?, sagt Damaris Bausch-Fluck, die Erstautorin nicht nur der obgenannten Studie ist, sondern auch von einem ?bersichtsartikel in der Fachzeitschrift ?Current Opinion in Chemical Biology?. Dort beschreibt sie zusammen mit den Kollegen, wie Oberfl?chenproteine im Nanometer Bereich organisiert sein k?nnten und wie diese Proteininseln die Funktion und Signalleitung beeinflussen koennen.
Folgende Analogie hilft, die Anordnung von Oberfl?chenproteinen und ihre Funktion zu verstehen: W?re ein Protein so gross wie ein Mensch, dann w?re die Fl?che der Zelloberfl?che dreimal so gross wie der Central Park in New York. Die Baseballfelder im Park w?ren dann eine solche funktionale Insel, auf der sich Proteine – die Spieler – zu einer besonderen Funktion – dem Baseballspiel – gruppieren. Verlassen die Spieler das Spielfeld, verlieren sie ihre Funktion.
Nachtr?gliche Ver?nderungen ver?ndern Funktion
Ausserdem stellt jeder Zelltyp die Oberfl?chenproteine in unterschiedlichen Konzentrationen her. Und schliesslich k?nnen Oberfl?chenproteine nach ihrer Herstellung ver?ndert, beispielsweise mit Zuckermolekülen oder Phosphaten ?dekoriert? werden. Dadurch übernehmen sie andere Funktionen als ohne diese molekulare Dekoration.
Bernd Wollscheid spricht daher nicht von Proteinen, sondern von Proteoformen. Das Grundgerüst mag immer gleich sein – durch die nachtr?glichen Ver?nderungen entstehen aus dem ursprünglichen Protein mehrere unterschiedliche Formen mit verschiedenen Funktionen.
Inseln als therapeutische Ziele
?Für die Entwicklung neuer Wirkstoffe ist es deshalb entscheidend, funktionale Einheiten mit mehreren Proteinen zu kennen und als m?gliche Zielstruktur ins Auge zu fassen?, sagt Wollscheid. Statt an ein spezifisches Protein k?nnten beispielsweise Antik?rper-Medikamente mehrere verschiedene Zielstrukturen gleichzeitig andocken, um eine funktionale Insel ausser Kraft zu setzen. Da die Inseln viel spezifischer sind für gewisse Zelltypen, etwa Krebszellen, ist es denkbar, dass sich Krankheiten auf diese Weise gezielter und mit weniger Nebenwirkungen behandeln lassen.
?Zurzeit sind wir daran, die vielen weissen Flecken auf den Oberfl?chen-Landkarten verschiedener Zelltypen zu erforschen – fast wie die Pioniere, die einst zu unbekannten Kontinenten aufbrachen?, erkl?rt Wollscheid.
Literaturhinweis
Bausch-Fluck D, Goldmann U, Müller S, van Oostrum M, Müller M, Schubert O, Wollscheid B. The in silico Human Surfaceome. PNAS November 13, 2018 115 (46) E10988-E10997; published ahead of print October 29, 2018 externe Seite doi:10.1073/pnas.1808790115
Bausch-Fluck D, Milani E, Wollscheid B. Surfaceome nanoscale organization and extracellular interaction networks. Current Opinion in Chemical Biology, Volume 48, February 2019, 26-33. externe Seite doi:10.1016/j.cbpa.2018.09.020